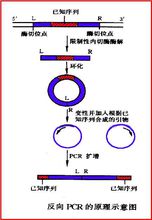
反向PCR
百科内容来自于:

原理
不足之处
其他用途
利用反向PCR可对未知序列扩增后进行分析,探索邻接已知DNA片段的序列,并可将仅知部分序列的全长cDNA进行分子克隆,建立全长的DNA探针。适用于基因游走、转位因子和已知序列DNA旁侧病毒整合位点分析等研究。
用传统的缓冲液和其他提供者推荐的条件裂解DNA。反向PCR所扩增的片段的大小由 PCR扩增片段的大小决定,目前,PCR扩增的实际上限为3-4kb。在许多情况下,首先 需要进行Southern杂交来确定内切酶用以产生大小适于环化及反向PCR的片段的末端 片段。能裂解核心区的内切酶使反向PCR只能扩增引物所定模板(依赖于引物)的上游 或上游区,而不裂解核心区的酶则使两上边侧序列都扩增,并带有由内切酶和环化类 型决定的接点(例如,互补突头连接与钝头连接)。对于扩增左翼或右翼序列,初试时 最好靠近识别上个碱基位位的酶,并已知在核心区有其方便的裂解位点。如果用反向 PCR从含有大量不同的克隆片段的同一载体中探测杂交探针,建议事先在载体中引入 合适的酶切位点。
$firstVoiceSent
- 来自原声例句